|
|
|
| 中國海洋大學在染色質解析與表觀遺傳編輯研究領域取得新進展 |
| http://www.gangguanzhidu.com 2025年12月10日 來源:華禹教育網 |
|
近日,中國海洋大學海洋生物多樣性與進化研究所原生動物學團隊高珊課題組在Cell Press細胞出版社旗下Trends in Genetics(《遺傳學趨勢》)發表綜述文章“Sequence-independent
6mA methyltransferases for epigenetic profiling and
editing”(無序列偏好性的6mA甲基化酶在表觀遺傳分析與基因編輯中的應用),系統梳理了外源6mA標記技術與三代長讀長測序相結合在染色質圖譜繪制、蛋白質—DNA互作定位和靶向表觀遺傳編輯方面的應用,著重分析了其方法學優勢,并就當前技術瓶頸和優化前景展開討論。
6mA作為一種新興的表觀遺傳標記,廣泛分布于原核生物和低等真核生物,在高等真核生物中含量稀少,這為利用6mA甲基化酶進行染色質標記提供了更低的背景。無序列偏好性的6mA甲基化酶EcoGII和Hia5催化活性高且底物范圍廣,已成為6mA表觀遺傳工程領域的核心工具。近年來,以Pacific Biosciences和Oxford Nanopore Technologies為代表的第三代測序技術,分別通過監測DNA聚合酶動力學擾動和離子電流變化,實現了對DNA甲基化修飾的單堿基分辨率直接檢測(圖1A)。相比二代測序,三代測序無需PCR擴增和DNA片段化處理,可直接檢測表觀遺傳標記并真實地反應單分子異質性,同時克服了二代測序在序列高度重復區域和結構復雜區域的多重比對問題(圖1B)。
通過將6mA甲基化酶和三代測序技術相結合,可實現單分子、單堿基分辨率下的染色質狀態解析,包括核小體占位情況、核小體缺失區域及轉錄因子結合位點的高精度檢測(圖2)。目前該技術已在玉米和人類腦組織等多個體系中得到了應用,雖仍受限于測序成本、細胞需求量和酶學條件等制約,但隨著測序價格下降以及酶工程和CRISPR富集技術發展,6mA甲基化酶有望成為繪制高分辨染色質圖譜和開展表觀診斷的重要工具。
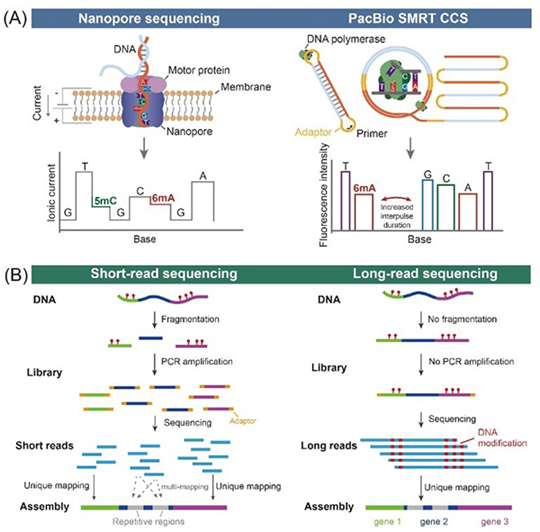
圖1 三代測序能夠在復雜區域進行DNA甲基化檢測
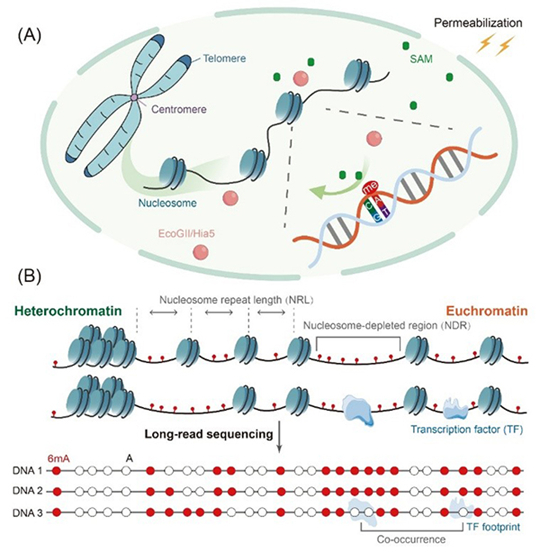
圖2 利用6mA甲基化酶和三代測序繪制染色質環境圖譜
在解析蛋白質—DNA相互作用方面,DiMeLo-seq等技術通過抗體識別目標蛋白,招募融合protein A的EcoGII或Hia5,在目標蛋白或組蛋白修飾位點附近特異性引入6mA標記,隨后借助三代測序技術,可直接在單分子水平獲得蛋白結合的位點信息,并可在序列高度重復的著絲粒和亞端粒區域檢測到新的蛋白互作位點。然而,抗體介導的非共價結合穩定性有限,后續可以引入納米抗體或采用共價偶聯策略增強復合物穩定性,從而進一步提升標記效率與位點解析的可靠性。
在靶向表觀基因編輯方面,將METTL4等6mA甲基化酶與dCas9融合表達,可在多種細胞中實現位點特異性6mA添加,再招募6mA識別蛋白與轉錄效應因子,能夠實現精確的轉錄調控。值得注意的是,EcoGII和Hia5由于其序列限制更弱,是構建6mA編輯工具的理想底盤,但需要通過載體設計、定向突變等方式優化其活性,從而降低脫靶效應。

高珊教授(后排左三)和論文第一作者張佳晨(后排左一)、張雨苗(前排右一)
該文章由中國海洋大學進化所原生動物學團隊高珊教授課題組撰寫。高珊教授課題組博士生張佳晨和張雨苗為該文章的共同第一作者,高珊教授為文章的通訊作者。高珊教授課題組博士生刁靜涵和美國南加州大學劉一凡副教授對本文亦有重要貢獻。研究工作得到國家自然科學基金、山東省自然科學基金、嶗山實驗室科技創新項目和中央高校基本科研業務費等資助。
文:張川
文章鏈接:https://www.sciencedirect.com/science/article/pii/S0168952525002367
|
 關于中國海洋大學更多的相關文章請點擊查看 關于中國海洋大學更多的相關文章請點擊查看  |
|
| 特別說明:由于各方面情況的不斷調整與變化,華禹教育網(www.gangguanzhidu.com)所提供的信息為非商業性的教育和科研之目的,并不意味著贊同其觀點或證實其內容的真實性,僅供參考,相關信息敬請以權威部門公布的正式信息為準。 |
|
|
|
| |